众所周知,目前市场上主要以二代测序为主,二代测序过程中第一步是文库构建,本期为大家介绍一下常见文库构建的种类。
针对于illumina测序平台,DNA类的文库主要分为:DNA小片段文库、DNA大片段文库、Exon文库、PCR-Free文库和单细胞文库等。
 DNA小片段建库
DNA小片段建库
DNA小片段文库是指片段大小在1kb以下的普通DNA文库(200bp、350bp、500bp等),DNA小片段文库可用来进行人重测序,动植物、微生物的de novo和重测序,16s rRNA测序,宏基因组测序等项目类型的文库构建。DNA小片段文库的构建主要包含以下2种方法:
01传统文库构建流程
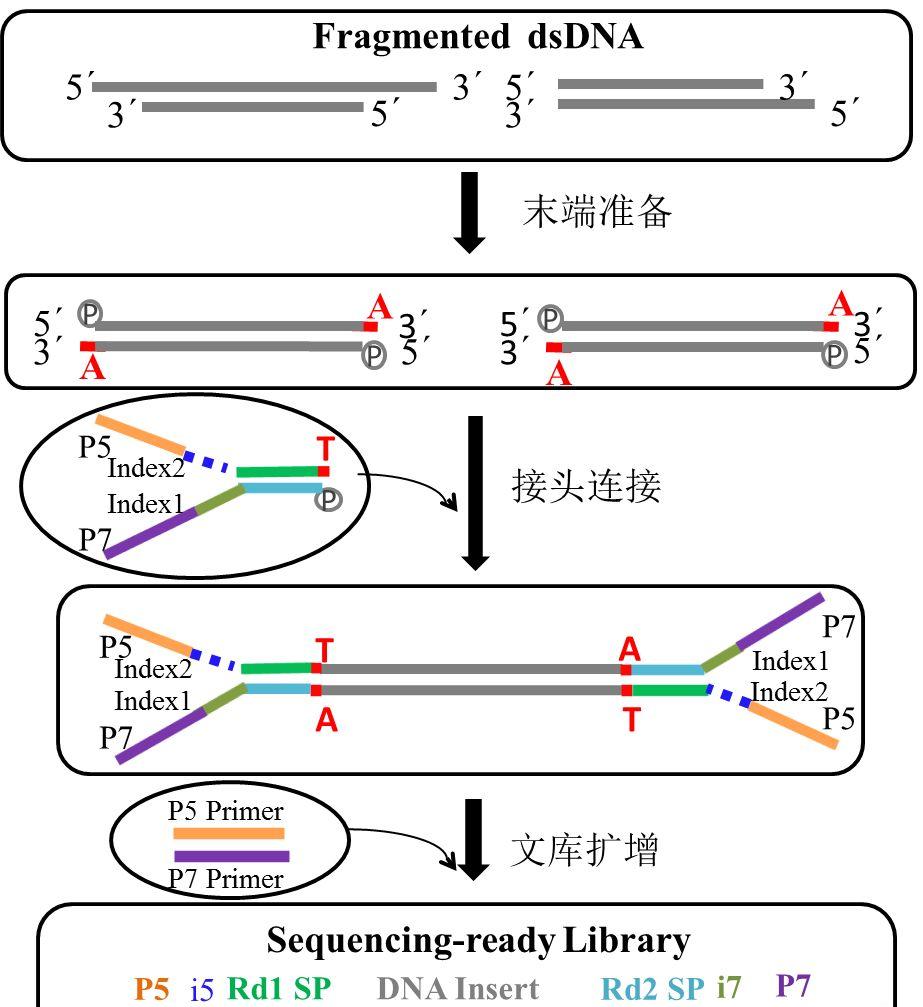
传统建库流程主要分为以下几个步骤:
(1)将待测序的DNA分子用超声波打碎成200-500bp长的序列片段
(2)将打碎后的基因组进行末端补平
(3)在补平后的片段3’端加A和5’端进行磷酸化
(4)对修复后的片段进行接头连接
(5)对接头连接后DNA片段进行PCR扩增
(6)对扩增后的文库进行磁珠纯化
DNA小片段文库建库原理图如下:
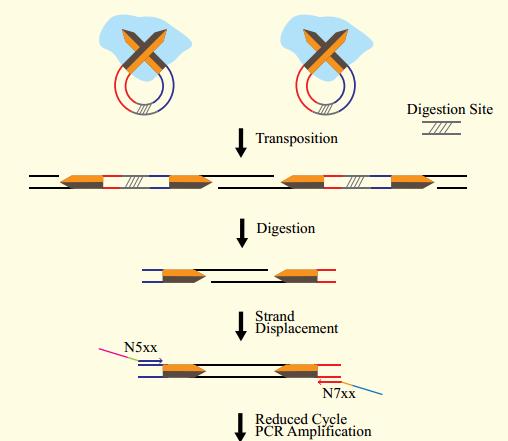
02转座法建库流程
转座法建库相对于传统建库,其实验方法简单快速,5分钟内即可同时完成DNA片段化和接头连接,而且所需要的DNA量少。目前市面上能提供具有自主知识产权专利的Tn5转座法建库试剂盒的公司就只有Illumina公司和TransGen公司。由于转座酶与所输入的基因组量有一定的比例关系,因此我司开发了2款转座酶法建库试剂盒:
(1)针对50ng起始量DNA
(2)针对1ng起始量DNA
转座法建库原理图如下:
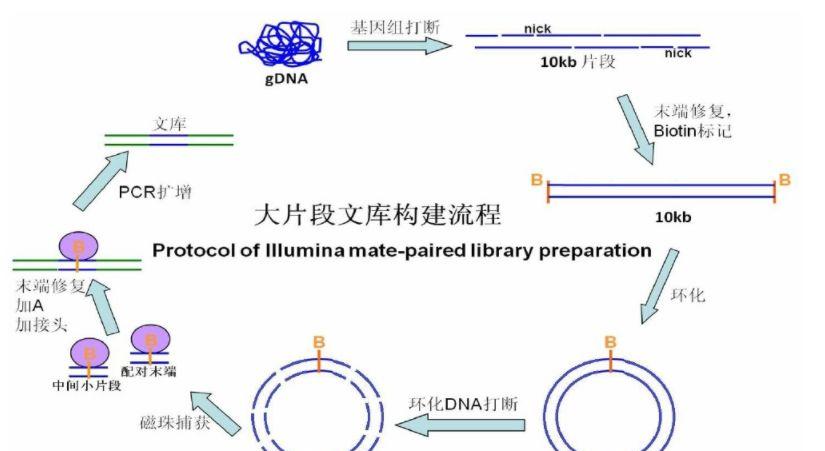
 DNA大片段建库
DNA大片段建库
DNA大片段文库,又称之为末端配对(mate-paired)文库,片段长度大于1kb,主要用于动植物,微生物的de novo测序。
DNA大片段文库的构建主要分为以下几个步骤:
(1)将待测序的基因组打断
(2)将打碎的基因组进行末端修复和进行Biotin标记
(3)将标记好的基因组环化
(4)将环化后的基因组打碎
(5)磁珠捕获片段化的基因组
(6)对捕获后的基因组进行末端修复加A和加接头
(7)对接头连接后的基因组进行PCR扩增
(8)将PCR扩增后的文库进行分选回收
DNA大片段文库建库流程图如下:
 Exon文库
Exon文库
人类外显子组总共约30Mb,占人类基因组约1%,外显子具有高度的保守型,且大部分疾病的致病位点位于外显子区。外显子测序是指利用序列捕获技术将外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。Exon文库主要用于人全外显子测序和目标区域测序。
外显子文库的构建流程主要分为以下几个步骤:
(1)将质量合格的基因组DNA超声打碎成200-300bp左右的片段
(2)将打碎的基因组DNA片段进行末端修复,3’端加A和5’端进行磷酸化
(3)对修复后的片段进行接头连接
(4)对接头连接后DNA片段进行线性扩增(LM-PCR)制备成杂交文库
(5)将构建好的文库进行探针杂交捕获
(6)将捕获后的文库进行PCR扩增富集
人全外显子测序与全基因组测序相比较,外显子测序具有测序覆盖度深,数据准确性高等优势,对于研究已知基因的SNP、Indel等具有较大的优势,因此主要应用在肿瘤基因测序方面。
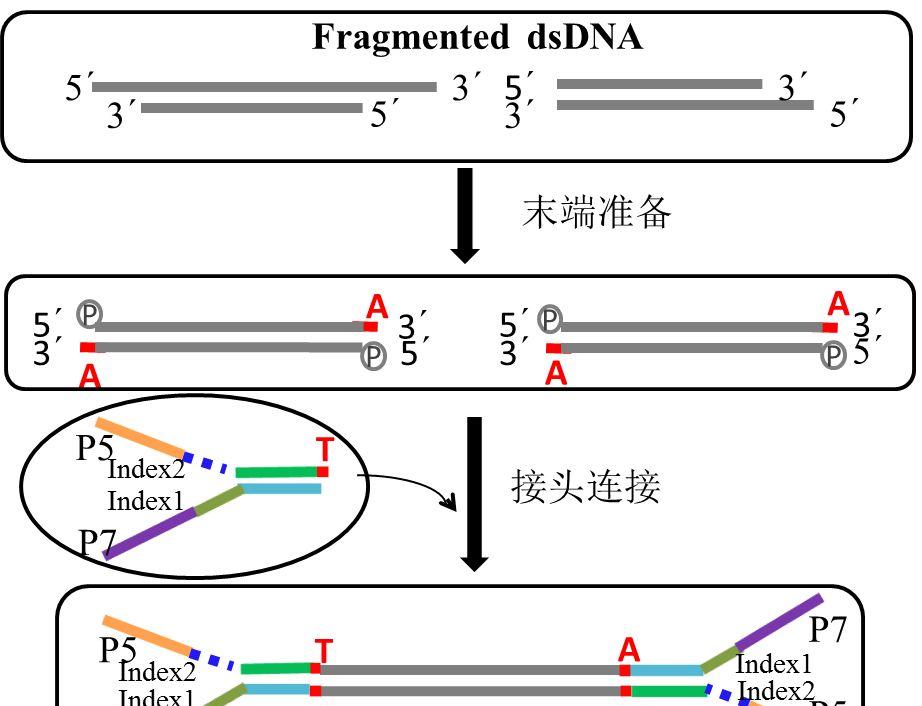
 PCR-Free文库
PCR-Free文库
PCR-Free文库,顾名思义,就是在文库构建过程中不需要进行PCR的文库。主要是针对一些特殊样本,比如GC含量高、PCR扩增困难的样本和PCR产物。不足之处在于所需的样本起始量较少。
PCR-Free文库的构建与传统小片段文库构建流程相比较,前面步骤相同,只是不需要进行PCR扩增富集。文库构建原理流程图如下:
 单细胞DNA文库
单细胞DNA文库
随着二代测序技术的发展,把一次实验测许多条DNA序列的这个难题解决之后,一次把一个人的全基因组给测出来,最极限的情况,就是样本量少到一个细胞,就可以测出整个基因组的序列信息。同时也需要克服以下三个难题:
(1)如何实现均匀扩增
(2)全基因组覆盖问题
(3)如何实现较高的扩增效率
为了解决上述的难题,科学家想了很多办法,到目前为止,大家比较认可的方法有2种:第一种是MALBAC方法,第二种是MDA方法。
1MALBAC方法
MALBAC方法,它的全称是:MultipleAnnealing and Looping-Based Amplification Cycles,是谢晓亮教授发明的方法,如下图所示:
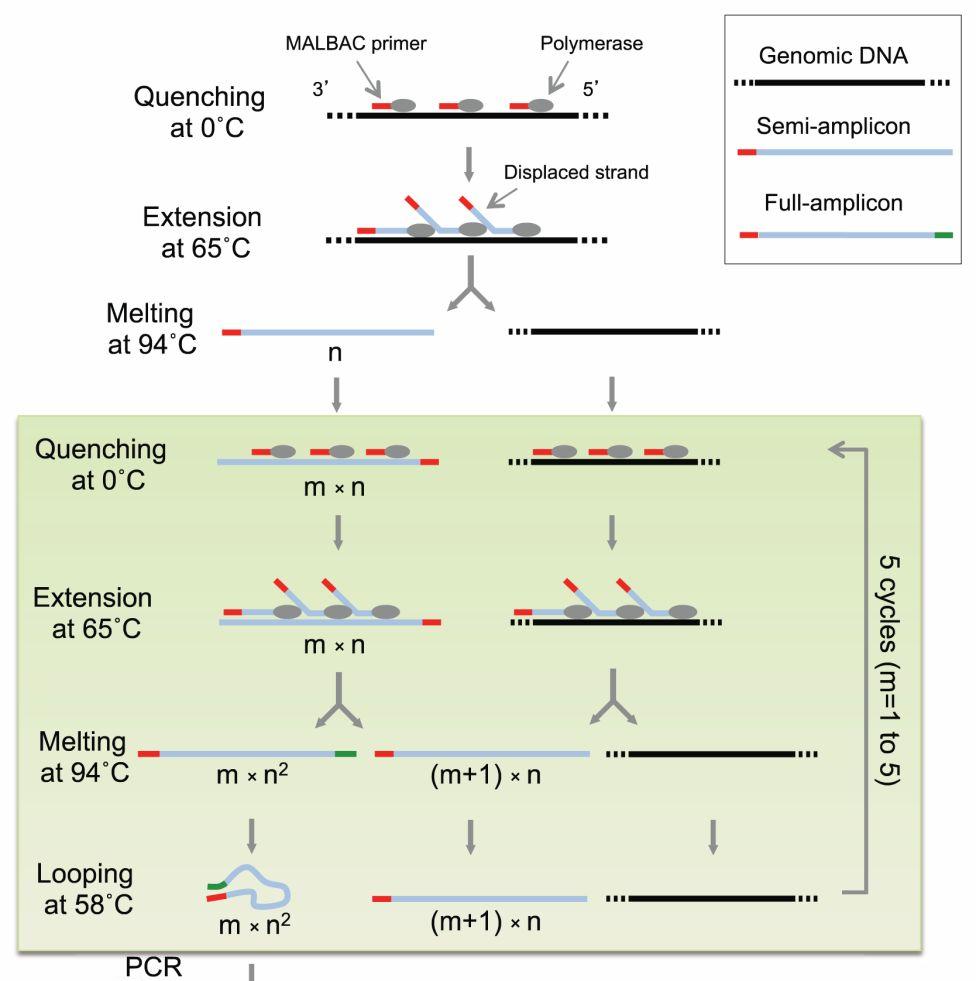
上图中黑色的线条,就是基因组模板DNA,这些红颜色的线条就是扩增引物,扩增引物的5’端有27个碱基的通用序列,这些通用序列会作为未来的PCR通用扩增引物的结合序列。扩增引物的3’端有8个随机序列的碱基,这8个碱基可以随机地杂交到基因组DNA的互补序列上。这些灰色的椭园是Phi 29 DNA聚合酶,Phi 29 DNA聚合酶有一个特点,它不仅可以生成新的DNA链,它还能把之前已经合成好的DNA链给解链开。再形成自己的新链,这个特点能够把每个循环所能合成的DNA新链的数量提高几倍、甚至几十倍、上百倍。接下来,就是做5个MALBAC循环,第一个循环得到的是 5’端有通用序列的DNA片段;第二个循环后,产生的扩增产物大部分是5’端有通用序列,3’端有与通用序列互补的序列片段。该方法在每个循环的最后,加了一步58度退火,这一退火过程让完整扩增的产物,它的两端发生链内杂交,这样3’端的序列就不能与新的、游离的引物发生杂交,也就不会引新的、发起始于3’端的扩增,就避免了完整扩整的产物的自我指数扩增。
MALBAC方法设计的扩增引物有8个随机序列,这样就可以在模板上随机地找结合位置,因此所有的位点都有一样的机会被扩增。该方法扩增的产物分为3种:第一种,就是m* n 个“完整扩增产物”,即最主要的产物,这里“m”就是循环的次数,“n”是一个循环中,有多少个扩增,引物可以粘到一个模板上。第2种扩增产物,就是(m 1)* n个“半扩增产物”,第3种DNA,就是原始的DNA模板,这里完整产物的数量是“m*n ”,也就是说,扩增产物(的数量)与扩增的循环次数“m”成正比,而不是与m的平方成正比,更不是与2 的M次方成正比,这就达到了我们想要的“线性扩增”的目的,即扩增产物(的数量)与扩增的次数成线性关系,进而实现了单细胞测序当中第一个要求“线性扩增”。第二个要解决的难题,就是“全基因组覆盖”问题,这里是利用Phi 29聚合酶一次在模板上能聚合出多个新链来达到这个目的。在5轮的扩增之后,每个模板都会有5*n^2个扩增片段,这样就可以保证建库时大多数的基因组区域可以被建成文库。 第三个要解决的问题“高效率扩增”,仍是利用了Phi 29酶一次能得到多个扩增片段来实现的。
2MDA方法
目前市场上还有一种单细胞扩增技术,叫MDA扩增技术,它的全称是Multiple Displacement Amplification。该方法的技术核心是利用Phi 29 DNA聚合酶来进行直接的扩增。Phi 29酶的特点是,它可以把双链DNA进行解链,然后在常温条件下,就可以将原始模板进行大量扩增。原理如下图所示:

上述两种方法各有优劣。MDA方法的优势在于实验方法简单,且扩增效率更高。MALBAC方法的特点在于它的扩增均一性更好,但是扩增效率相对比较低。
目前单细胞测序技术的应用主要有以下几个方面:
(1)体外受精胚胎测序
(2)CTC测序,找到突变位点,针对性地进行靶向药物治疗
(3)肿瘤亚细胞群测序,监测肿瘤细胞不断发生的新的突变,并找到对应的治疗方案
(4)免疫细胞测序,免疫细胞在对抗原发生识别后,其基因组发生重排并产生对抗原特异的抗体,测序可找出特异的基因结构
以上为本期为您介绍的内容,希望对您的科研有所帮助,下期见!
研发部 白 霞 ▏文案